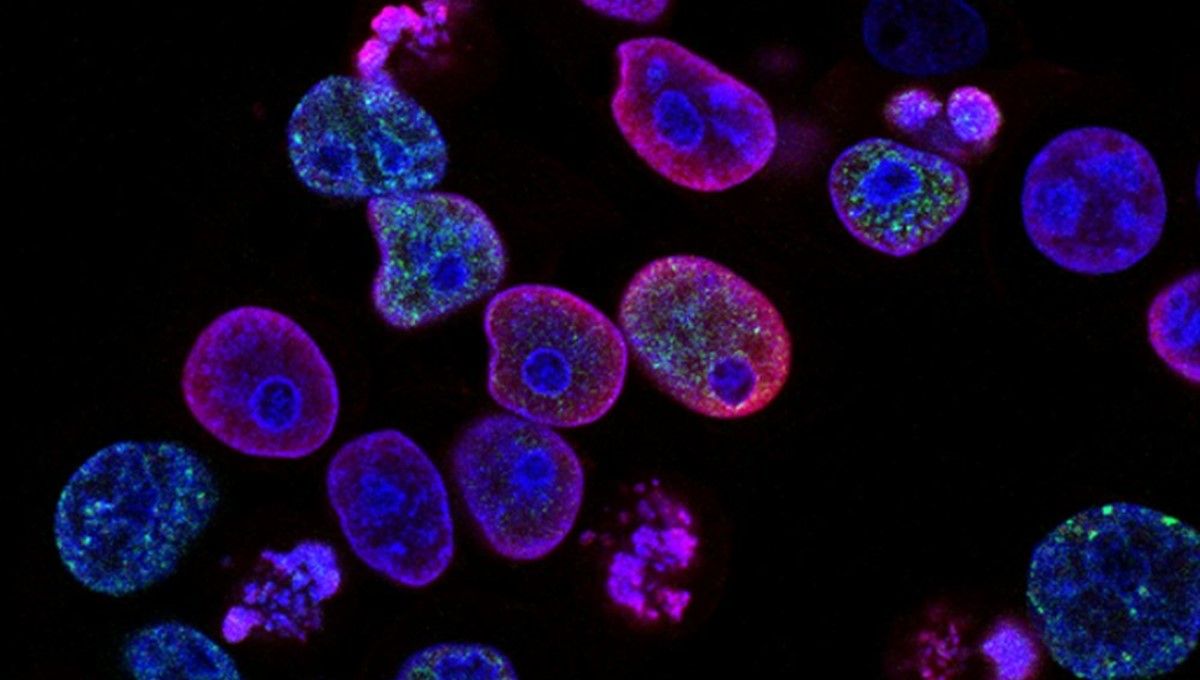
Investigadores de la Universidad de Pekín han diseñado un nuevo método destinado a mapear células que supera las limitaciones de los anteriores sistemas. Se trata del método 3DOM, que ha arrojado nueva información sobre la orientación dipolar de los orgánulos de células vivas, así como de la variedad de estructuras que componen estas células y sus orgánulos.
Las células presentan una alta transparencia, por lo que es muy complicado llegar a observar los orgánulos que contienen. Hasta ahora, los investigadores utilizaban métodos convencionales mediante tinción fluorescente, una técnica que aplicaba fluorescencia a orgánulos específicos para que los expertos pudieran etiquetarlas y trabajar con ellas. Sin embargo, uno de los desafíos de la tinción fluorescente era resolver la incógnita de la información angular u orientación.
La mayoría de las moléculas fluorescentes aparecen como dipolos direccionales durante la absorción o la emisión, pero se sabe que la orientación de los fluoróforos puede revelar información importante sobre la estructura y dinámica de sus orgánulos asociados. Ahora ese reto parece quedar resuelto, después de que se hayan propuesto técnicas mejoradas de microscopía de polarización de fluorescencia de superresolución.
Uno de los desafíos de la tinción fluorescente era resolver la incógnita de la información angular u orientación
Las técnicas convencionales de mapeo de células incluyen la microscopía de orientación-localización de una sola molécula (SMOLM) y la modulación de polarización, como los métodos, SDOM o SPoD. Ahora, el equipo de investigación del profesor Xi Peng de la Universidad de Pekín ha presentado un microscopio de mapeo de orientación 3D (3DOM) de superresolución, que logra una precisión azimutal de 2° y una precisión polar de 3° con una resolución espacial de hasta 128 nm. Este modelo se sirve del desarrollo de un software de código abierto y un hardware de código abierto para el análisis.
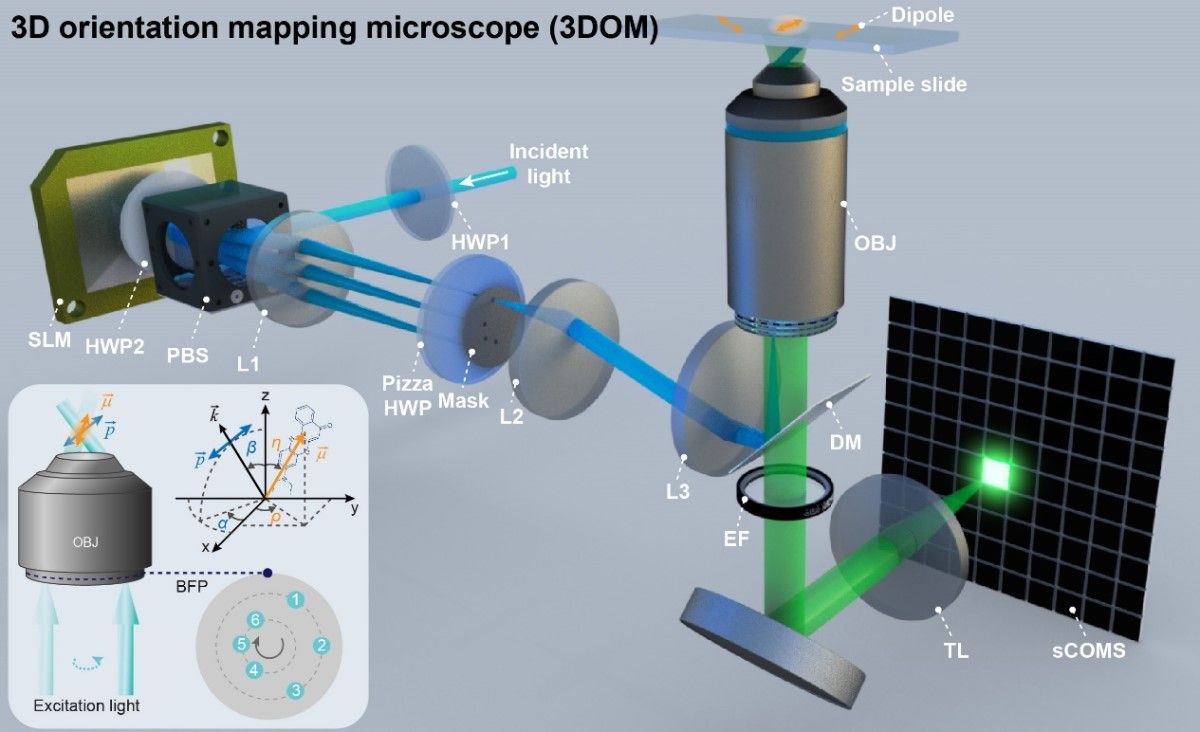
El sistema resuelve el problema de la orientación dipolar a través de la iluminación estructurada polarizada desarrollada por el propio equipo de científicos. Para este punto se utilizan diferentes ángulos de las franjas para producir haces de luz de primer orden positivos y negativos en diferentes direcciones, todo ello invirtiendo el principio de interferencia de doble rendija y combinándolo con el principio de trayectorias de luz reversibles.
Este proceso permite generar una única orientación de iluminación inclinada mediante el bloqueo directo de la luz negativa de primer orden correspondiente. Al aplicar esta inclinación a distintos ángulos del eje Z y reconstruir la imagen mediante el algoritmo FISTA, se puede alcanzar una resolución de alta precisión en la orientación del dipolo al fusionar los coeficientes de modulación de polarización con los resultados de la reconstrucción en el espacio recíproco.
3DOM ofrece una perspectiva más detallada de la disposición tridimensional espacial de las moléculas de fluoróforo. Esto posibilita distinguir distintas organizaciones citoesqueléticas, como los filamentos de actina y microtúbulos, y obtener datos valiosos sobre la compactación de los filamentos y la disposición de las estructuras subcelulares. Además, 3DOM presenta un potencial significativo en la curvatura del ADN y la alineación de orgánulos membranosos.
El sistema 3DOM permitirá a los distintos investigadores arrojar más luz sobre la composición de la estructura subcelular y los procesos celulares
Tras los experimentos, el nuevo método también ha revelado la heterogeneidad de la membrana del glóbulo de grasa láctea. Asimismo, ha obtenido imágenes de la orientación dipolar de los microtúbulos marcados con proteína verde fluorescente, informando así de las variaciones dinámicas de orientación 3D. Este era el reto mayor al que se enfrentaba el proyecto y que ha superado con éxito.
Una de las ventajas principales de 3DOM es que ofrece una amplia gama de aplicaciones y adaptabilidad a los sistemas de campo amplio ya existentes, mejorando así la accesibilidad y usabilidad de los mapeos celulares convencionales. Además, permitirá a los distintos investigadores arrojar más luz sobre la composición de la estructura subcelular y los procesos celulares. El éxito del proyecto también ha abierto nuevos horizontes a sus autores, quienes se han comprometido a desarrollar nuevos tipos de tecnología de microscopía de superresolución.