La información en la secuencia del genoma humano tiene una importancia primordial en la investigación biomédica. Sin embargo, el valor de esta información es muy limitado, puesto que falta un mapa detallado de los genes codificados en el genoma. Los genes son las unidades biológicas básicas responsables de los rasgos biológicos del organismo. Ya existe información detallada sobre las regiones genómicas que contienen los genes que codifican las proteínas, pero la información sobre las regiones de ADN no codificantes, también conocidas como ADN "materia oscura", no está avanzado. Es donde se encuentran genes poco conocidos llamados "ARN largos no codificantes" (lncRNA), que se encuentran entre los más numerosos de todos, y se han relacionado con una variedad de enfermedades.
En un artículo publicado en Nature Genetics, un equipo internacional de científicos dirigido por investigadores del Centro de Regulación Genómica (CRG) en Barcelona, en colaboración con investigadores de Cold Spring Harbor en Nueva York, el Wellcome Trust Sanger Institute en Hinxton, y qGenomics en Barcelona arroja luz sobre este tema. Para identificar, mapear y caracterizar mejor esos genes de "materia oscura", han desarrollado un nuevo método que mejora el rendimiento y la precisión de los métodos actuales, y lo aplica en humanos y ratones.
"El 98% de nuestro ADN no codifica para proteínas. Estas regiones de ADN contienen miles de genes no codificantes que no están caracterizados y todavía queda un largo camino hasta que comprendamos sus funciones y su papel en las enfermedades"
"El 98% de nuestro ADN no codifica para proteínas. Estas regiones de ADN contienen miles de genes no codificantes que no están caracterizados y todavía queda un largo camino hasta que comprendamos sus funciones y su papel en las enfermedades. Para ello, necesitamos tener mapas completos de todos los genes. Nuestro método representa un paso muy importante en esta dirección", explica Rory Johnson, antiguo investigador en el CRG que actualmente es jefe de grupo en la Universidad de Berna, y codirige de este trabajo.
La principal característica de este nuevo método, llamado RNA Capture Long Seq (CLS), es que se centra específicamente en las regiones no codificantes del genoma. Éstas se han amplificado y analizado mediante las técnicas más avanzadas de secuenciación. "De este modo, hemos podido producir un mapa detallado de más de 3.500 ARN largos no codificantes en humanos y ratones. Y eso nos ha permitido describir las características genómicas de ARN largos no codificantes para comprender mejor cómo funcionan", comentan los investigadores Julien Lagarde y Barbara Uszczynska, primeros autores del trabajo en el CRG.
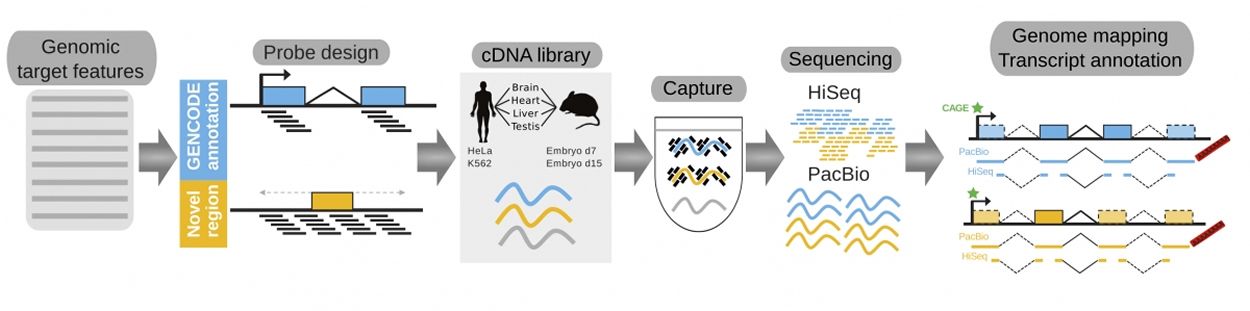
Los investigadores usaron este nuevo método para mejorar una de las bases de datos genómicas más importantes: Gencode, que es la referencia mundial para los genes codificantes en los genomas de humano y ratón. "Científicos en todo el mundo están utilizando Gencode para sus proyectos de investigación como datos de referencia. Así que mejorar Gencode, implica contribuir a la investigación biomédica mundial", afirma Roderic Guigó, coordinador del programa de Bioinformática y Genómica en el CRG y colíder del trabajo. Guigó inició Gencode en 2003 como parte del proyecto Encode La Enciclopedia de los Elementos del ADN. Ahora, gracias a este nuevo método, Guigó y colaboradores han mejorado sustancialmente los catálogos de genes, en concreto, los genes de ARN largos no codificantes. "Hemos encontrado un método más rápido, preciso y económico que nos ha permitido mejorar un catálogo que beneficiará en un primer momento a la comunidad científica, para luego, beneficiar a toda la sociedad", concluye Guigó.

